蛋白質結構

蛋白質結構是指蛋白質分子的空間結構。作為一類重要的生物大分子,蛋白質主要由碳、氫、氧、氮、硫等化學元素組成。絕大部分蛋白質都是由20種不同的L型α胺基酸連接形成的聚合物,但還存在另外兩種不常見胺基酸,硒半胱胺酸、吡咯離胺酸,參與到極少數的蛋白質合成中(其轉譯機制也不同於61種密碼子)。在形成蛋白質後,這些胺基酸又被稱為殘基。
蛋白質和多肽之間的界限並不是很清晰,有人基於發揮功能性作用的結構域所需的殘基數認為,若殘基數少於40,就稱之為多肽或肽。
要發揮生物學功能,蛋白質需要正確摺疊為一個特定構型,主要是透過大量的非共價相互作用(如氫鍵、離子鍵、范德華力和疏水作用)來實現;此外,在一些蛋白質(特別是分泌性蛋白質)摺疊中,雙硫鍵也起到關鍵作用。為了從分子水平上了解蛋白質的作用機制,常常需要測定蛋白質的三維結構。由研究蛋白質結構而發展起來了結構生物學,採用了包括X射線晶體學、核磁共振以及冷凍電鏡(cryo-EM)等技術來解析蛋白質結構。
一定數量的殘基對於發揮某一生物化學功能是必要的;40-50個殘基通常是一個功能性結構域大小的下限。蛋白質大小的範圍可以從這樣一個下限一直到數千個殘基。目前估計的蛋白質的平均長度在不同的物種中有所區別,一般約為200-380個殘基,而真核生物的蛋白質平均長度比原核生物長約55%。[1]更大的蛋白質聚合體可以透過許多蛋白質亞基形成;如由數千個肌動蛋白分子聚合形成蛋白纖維。



不同層次的蛋白質結構
[編輯]
蛋白質的分子結構可劃分為四級,以描述其不同的方面:
- 蛋白質一級結構:組成蛋白質多肽鏈的線性胺基酸序列。一個蛋白質是一個聚醯胺。
- 蛋白質二級結構:依靠不同胺基酸之間的C=O和N-H基團間的氫鍵形成的穩定結構,主要為α螺旋和β摺疊。
- 蛋白質三級結構:透過多個二級結構元素在三維空間的排列所形成的一個蛋白質分子的三維結構。
- 蛋白質四級結構:用於描述由不同多肽鏈(亞基)間相互作用形成具有功能的蛋白質複合物分子。
除了這些結構層次,蛋白質可以在多個類似結構中轉換,以行使其生物學功能。對於功能性的結構變化,這些三級或四級結構通常用化學構象進行描述,而相應的結構轉換就被稱為構象變化。
一級結構是透過共價鍵(肽鍵)形成。生物體中,肽鍵的形成是發生在蛋白質生物合成的轉譯步驟。胺基酸鏈的兩端,根據末端自由基團的成分,分別以「N末端」(或「胺基端」)和「C末端」(或「羧基端」)來表示。
定義不同類型的二級結構有不同的方法,[2][3][4]最常用的方法是透過主鏈原子之間的氫鍵的排列方式來判斷的。而在蛋白質完全摺疊的狀態下,這些氫鍵可以得到穩定。
三級結構主要是透過結構「非特異性」相互作用來形成。然而,只有當蛋白質結構域透過「特異性」相互作用(如鹽橋,氫鍵以及側鏈間的堆積作用)固定到相應位置,所形成的三級結構才能穩定。對於細胞外周蛋白,二硫鍵起到了關鍵的穩定作用;而對於細胞內蛋白質,則很少出現二硫鍵,因為原生質中是還原環境,不利於二硫鍵的形成。[5]
胺基酸結構
[編輯]

α-胺基酸由一個所有胺基酸類型中都含有的共同部分(形成蛋白質的主鏈)和一個對每一類胺基酸都不同的側鏈所組成。如右圖所示,「Cα」原子連接著4個不同類別的原子或基團:一個胺基、一個羧基、一個氫原子(圖中略去氫原子)和一個條側鏈(用「R」表示,以代表各種不同的胺基酸的側鏈)。不完全符合這一特性的一個特例是脯胺酸,其Cα原子沒有連接氫原子而是被側鏈取代。由於連接著不同的4個基團,這就使胺基酸有了手性;但大多數蛋白質都是同一構型的(左手型的同手性)。由於甘胺酸沒有側鏈(或者說側鏈為一個氫原子),因此沒有手性。左手型的胺基酸可以用一個簡單的「CORN」法則來記憶:以氫原子在前來看Cα原子,其他三個基團「CO-R-N」以順時針方向排布。 側鏈決定了20種α-胺基酸的化學性質,具體如下表:
| 殘基名稱 | 三字母 代碼 |
單字母 代碼 |
相對豐度 (%) E.C. |
分子量 | pKa[6] | VdW體積 (ų) |
帶電(C), 極性(P), 疏水性(H) |
|---|---|---|---|---|---|---|---|
| 丙胺酸(Alanine) | ALA | A | 13.0 | 71 | 67 | H | |
| 精胺酸(Arginine) | ARG | R | 5.3 | 157 | 12.5 | 148 | C+ |
| 天門冬醯胺(Asparagine) | ASN | N | 9.9 | 114 | 96 | P | |
| 天門冬胺酸(Aspartate) | ASP | D | 9.9 | 114 | 4.5 | 91 | C- |
| 半胱胺酸(Cysteine) | CYS | C | 1.8 | 103 | 8.3 | 86 | P |
| 麩胺酸(Glutamate) | GLU | E | 10.8 | 128 | 4.5 | 109 | C- |
| 麩醯胺酸(Glutamine) | GLN | Q | 10.8 | 128 | 114 | P | |
| 甘胺酸(Glycine) | GLY | G | 7.8 | 57 | 48 | ||
| 組胺酸(Histidine) | HIS | H | 0.7 | 137 | 6.8 | 118 | P,C+ |
| 異白胺酸(Isoleucine) | ILE | I | 4.4 | 113 | 124 | H | |
| 白胺酸(Leucine) | LEU | L | 7.8 | 113 | 124 | H | |
| 離胺酸(Lysine) | LYS | K | 7.0 | 129 | 11.1 | 135 | C+ |
| 甲硫胺酸(Methionine) | MET | M | 3.8 | 131 | 124 | H | |
| 苯丙胺酸(Phenylalanine) | PHE | F | 3.3 | 147 | 135 | H | |
| 脯胺酸(Proline) | PRO | P | 4.6 | 97 | 90 | H | |
| 絲胺酸(Serine) | SER | S | 6.0 | 87 | 73 | P | |
| 蘇胺酸(Threonine) | THR | T | 4.6 | 101 | 93 | P | |
| 色胺酸(Tryptophan) | TRP | W | 1.0 | 186 | 163 | P | |
| 酪胺酸(Tyrosine) | TYR | Y | 2.2 | 163 | 9.8 | 141 | P |
| 纈胺酸(Valine) | VAL | V | 6.0 | 99 | 105 | H |
基於化學性質的不同,可以將20種天然胺基酸分成多個類別。重要的影響因子是側鏈帶電性、親/疏水性、大小等。不同側鏈在水溶液環境中的相互作用在塑造和維持蛋白質結構中扮演著重要的角色。疏水性的側鏈趨向於被包埋於蛋白質內部,形成疏水核心,穩定蛋白質結構;而親水性的側鏈則更多的是暴露於溶劑中。疏水性的殘基包括白胺酸、異白胺酸、苯丙胺酸和纈胺酸以及疏水性相對較弱的甘胺酸、丙胺酸、色胺酸和甲硫胺酸。帶電側鏈對於蛋白質結構的穩定性也非常重要,透過不同帶電側鏈之間形成離子鍵可以穩定結構,而如果結構內部有未配對的帶電側鏈則會大大減弱結構的穩定性;此外,帶電殘基有很強的親水性,通常位於蛋白質表面。帶正電的殘基有離胺酸和精胺酸,有時組胺酸也帶正電荷;帶負電的殘基為麩胺酸和天門冬胺酸。其餘的胺基酸一般有帶不同功能基團的較小的親水側鏈。如絲胺酸和蘇胺酸側鏈帶羥基,麩醯胺酸和天門冬醯胺帶醯胺基。一些胺基酸具有特殊性質,如兩個半胱胺酸之間能夠透過側鏈上的巰基共價連接而形成二硫鍵,脯胺酸為環狀且構象比較固定,甘胺酸為最小胺基酸且構象最具可變性。
肽鍵
[編輯]

兩個胺基酸可以透過縮合反應結合在一起,並在兩個胺基酸之間形成肽鍵。而不斷地重複這一反應就可以形成一條很長的殘基鏈(即多肽鏈)。這一反應是由核糖體在轉譯進程中所催化的。肽鍵雖然是單鍵,但具有部分的雙鍵性質(由C=O雙鍵中的π電子云與N原子上的未共用電子對發生共振導致),因此C-N鍵(即肽鍵)不能旋轉,從而連接在肽鍵兩端的基團處於一個平面上,這一平面就被稱為肽平面。而對應的肽二面角φ(肽平面繞N-Cα鍵的旋轉角)和ψ(肽平面繞Cα-C1鍵的旋轉角)有一定的取值範圍;一旦所有殘基的二面角確定下來,蛋白質的主鏈構象也就隨之確定。根據每個殘基的φ和ψ來做圖,就可以得到拉氏圖,由於形成同一類二級結構的殘基的二面角的值都限定在一定範圍內,因此在拉氏圖上就可以大致分辨殘基參與形成哪一類二級結構。下表列出了肽鍵與對應類型單鍵以及氫鍵鍵長的比較。
| 肽鍵 | 平均長度 | 單鍵 | 平均長度 | 氫鍵 | 平均長度(±30) |
|---|---|---|---|---|---|
| Cα - C | 153 pm | C - C | 154 pm | O-H --- O-H | 280 pm |
| C - N | 133 pm | C - N | 148 pm | N-H --- O=C | 290 pm |
| N - Ca | 146 pm | C - O | 143 pm | O-H --- O=C | 280 pm |
一級結構
[編輯]肽或蛋白質的胺基酸序列(或殘基序列)被稱為蛋白質一級結構。殘基的標號總是從蛋白質的胺基端(沒有參與形成肽鍵)開始。蛋白質一級結構可以透過測定其對應的基因(更準確地說是開放閱讀框架)的鹼基序列來間接確定(參見轉譯),但對於轉錄後修飾和轉譯後修飾,如二硫鍵形成、磷酸化和醣基化等(通常被認為是一級結構的組成資訊),則無法透過這種轉譯法來測定;此外,也可以透過埃德曼降解法或連續質譜來對蛋白質樣品進行直接定序。

二級結構
[編輯]
早在1951年,第一個蛋白質結構解出前7年,鮑林和他的同事就利用已知的鍵長和鍵角提出了α螺旋和β摺疊的結構。[7]α螺旋和β摺疊都是將主鏈上的氫鍵供體和受體飽和的一種方式。這兩個蛋白質二級結構僅依賴於主鏈骨架,即所有胺基酸的共同部分,這就解釋了為什麼這兩個蛋白質二級結構頻繁地出現於大多數的蛋白質結構中。隨著越來越多的蛋白質結構得到解析,更多的蛋白質二級結構被發現,如各類Loop和其他形式的螺旋。蛋白質二級結構都有自己獨特的幾何構架,即二面角ψ和φ有特定的值,處於Ramachandran圖的特定區域。蛋白質二級結構還包括轉角、Loop和其他一些不常見的二級結構元素(如310螺旋等)。除了有規則的二級結構以外,主鏈骨架的其他部分就被稱為無規則捲曲。
 |
 |
 |
三級結構
[編輯]蛋白質二級結構元素通常被摺疊為一個緊密形態,元素之間以各種類型的環(loop)和轉角相連。蛋白質三級結構的形成驅動力通常是疏水殘基的包埋,但其他相互作用,如氫鍵、離子鍵和二硫鍵等同樣也可以穩定三級結構。蛋白質三級結構包括所有的非共價相互作用(不包括二級結構),並定義了蛋白質的整體摺疊,對於蛋白質功能來說是至關重要的。
四級結構
[編輯]蛋白質四級結構是由兩個或多個多肽鏈透過相互作用形成的結構。其中,單獨的一條鏈就被稱為亞基。不是所有的蛋白質都有四級結構,許多蛋白可以以單體形式來發揮功能。蛋白質四級結構的穩定性與其三級結構處於同一水平。兩個或多個亞基形成的複合物統稱為多聚體(multimer),如果是兩個亞基則稱二聚體或二體(dimer),三個亞基稱三聚體或三體(trimer),以此類推。如果多聚體為相同的亞基組成,則加上「同源(homo-)」作為前綴,反之則用「異源(hetero-)」,如同源二聚體或異源三聚體。
側鏈構象
[編輯]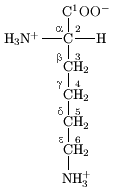
殘基側鏈上的原子根據希臘字母表的順序(α、β、γ、δ、ε等)來命名,如Cα指的是對應殘基上最接近羰基的碳原子,而Cβ則是次接近的。Cα通常被認為是主鏈骨架的組成原子。這些原子之間的鍵對應的二面角則相應以χ1、χ2、χ3等來命名,如離胺酸側鏈上第一、二個碳原子(即Cα和Cβ)之間共價鍵的二面角為χ1。側鏈可以有多種不同的構象,每一種類型的殘基都有幾種比較穩定的側鏈構象。[8]
結構域、結構模體與摺疊類型
[編輯]
蛋白質經常描述為由幾個結構單元所構成。這些結構單位包括結構域,模體,和摺疊。儘管真核生物體可以表現數萬種不同的蛋白質,但對應的結構域、模體與摺疊類型的數量卻少得多。一種合理的解釋是,這是進化的結果;因為基因或基因的一部分可以在基因組內被加倍或移動。也就是說,透過基因重組,一個結構域可以從相應蛋白質A移動到本不具有此結構域的蛋白質B上,而其發生的進化驅動力可能是由於該結構域對應的生物學功能趨向於被蛋白質B所利用。
蛋白質結構域
[編輯]許多蛋白質都可以被分為多個結構組成單元,蛋白質結構域(Protein domains)就是這樣一個組成單元。結構域一般可以自穩定,且常常獨立進行蛋白質摺疊,而不需要蛋白質其他部分的參與;很多結構域都有自己獨特的生物學功能。很多結構域並不是一個基因或基因家族對應蛋白質的獨特結構單元,而往往是許多類蛋白質的共同結構單元。因為它們所屬的蛋白質的生物學功能中占據顯著地位,蛋白質結構域通常被命名和被挑選出來; 例如,「鈣調蛋白的鈣結合結構域」。或者以幾類最初發現此結構域的蛋白名稱衍生而來,例如PDZ結構域(最初發現於PSD95、DlgA和ZO-1這三個蛋白質)。由於結構域自身可以穩定存在,因此可以將不同來源的結構域透過基因工程人為地結合在一起,形成融合蛋白質。
結構模體和序列模體
[編輯]結構模體(structural motif)和序列模體是指在大量不同蛋白質中被發現的蛋白質的三維結構或胺基酸序列的短的片斷。結構模體是一種結構組成單元,它是由幾個二級結構的特定組合(如螺旋-轉角-螺旋)所組成;這些組合又被稱為超二級結構。結構模體往往還包含有長度不同的loop區。
蛋白質摺疊
[編輯]摺疊類型則指的是整體的結構排列類型,如螺旋束、β桶、和羅斯曼摺疊,或是由蛋白質資料庫結構分類中提供的不同的摺疊。[9]
從一級結構到更高級結構的過程就被稱為蛋白質摺疊。一個序列特定的多肽鏈(摺疊之前的蛋白質一般都被稱為多肽鏈)一般摺疊為一種特定構象(又稱為天然構象);但有時可以摺疊為一種以上的構象,且這些不同構象具有不同的生物學活性。在真核細胞內,許多蛋白質的正確摺疊需要分子伴侶的幫助。

結構分類
[編輯]對蛋白質結構進行分類的方法有多種,有多個結構資料庫(包括SCOP、CATH、和FSSP)資料庫提供不同的結構分類。分別採用不同的方法進行結構分類。存放蛋白質結構的PDB資料庫中就引用了SCOP的分類。對於大多數已分類的蛋白質結構來說,SCOP、CATH和FSSP的分類是相同的,但在一些結構中還有所區別。
結構測定
[編輯]

在專門存儲蛋白質和核酸分子結構的蛋白質資料庫中,接近90%的蛋白質結構是用X射線晶體學的方法測定的[10]。X射線晶體學可以藉由測定蛋白質分子在晶體中電子密度的空間分布,在一定解析度下解析蛋白質中所有原子的三維坐標。大約9%的已知蛋白質結構是經由核磁共振技術測定[10]。該技術還可用於測定蛋白質的二級結構。蛋白質二級結構的組成可以透過圓二色性測定。振動光譜法也可用於表徵肽,多肽和蛋白質的構象[11] 。二維紅外光譜已成為研究不能用其他方法研究的柔性肽和蛋白質的結構的有價值的方法[12][13]。低溫電子顯微鏡(Cryo-electron microscopy)是近年來興起的一種獲得低解析度(低於5埃)蛋白質結構的方法,該方法最大的優點是適用於大型蛋白質複合物(如病毒外殼蛋白、核糖體和類澱粉蛋白纖維)的結構測定;並且在一些情況下也可獲得較高解析度的結構,如具有高對稱性的病毒外殼和膜蛋白二維晶體[14][15]。
近年來,隨著結構基因組學的興起,大量的蛋白質結構獲得了測定,為研究蛋白質的作用機理提供了重要的結構資訊。
結構預測
[編輯]測定蛋白質序列比測定蛋白質結構容易得多,而蛋白質結構可以給出比序列多得多的關於其功能機制的資訊。因此,許多方法被用於從序列預測結構。
- 二級結構預測
- 三級結構預測
- 同源建模:需要有同源的蛋白三級結構為基礎進行預測。
- Threading法。
- 「從頭開始」(Ab initio):只需要蛋白質序列即可進行結構預測。由於運算量大,需要有超級計算機來進行,或採用分布式計算,如Rosetta@home等。
- 四級結構預測:主要是預測蛋白質-蛋白質之間的相互作用方式。
相關軟體
[編輯]與蛋白質結構相關的軟體有很多,主要分為以下幾類:
- 三維結構圖形化顯示。較為流行的有PyMOL、Rasmol、MolMol等。
- 三維結構解析。包括晶體結構解析、NMR結構解析和電鏡結構解析。著名的軟體包有CCP4和CNS等。
- 結構預測:
- 結構分析。這一類軟體數量龐大,功能不同,各有特色,以下列出其中較為常用的一些功能和對應軟體:
更多軟體可以在ExPASy Proteomics tools (頁面存檔備份,存於網際網路檔案館)上查找。
參閱
[編輯]參考文獻
[編輯]- ^ (英文)Brocchieri L and Karlin S. Protein length in eukaryotic and prokaryotic proteomes. Nucleic Acids Res. 2005, 33: 3390–3400. PMID 15951512.
- ^ (英文)Kabsch, W and Sander, C. A dictionary of protein secondary structure. Biopolymers. 1983, 22: 2577–2637.
- ^ (英文)Richards, FM and Kundrot, CE. Identification of structural motifs from protein coordinate data: secondary structure and first-level supersecondary structure. Proteins. 1988, 3: 71–84.
- ^ (英文)Frishman, D and Argos, P. Knowledge-based protein secondary structure assignment. Proteins. 1995, 23: 566–579.
- ^ (英文)Freedman RB, Hirst TR, Tuite MF. Protein disulphide isomerase: building bridges in protein folding. Trends Biochem Sci. 1994, 19: 331–336.
- ^ (英文)Amino Acid pKa values (頁面存檔備份,存於網際網路檔案館),表中顯示的為胺基酸側鏈的pKa值。
- ^ (英文)PAULING L, COREY RB, BRANSON HR. Proc Natl Acad Sci U S A. 1951 Apr;37(4):205-11. The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain. PMID 14816373
- ^ (英文)Protein Sidechain Conformational Analysis. [2008年1月30日]. (原始內容存檔於2008年2月24日).
- ^ Govindarajan S, Recabarren R, Goldstein RA. Estimating the total number of protein folds.. Proteins. 17 September 1999, 35 (4): 408–414 [2015-02-28]. PMID 10382668. doi:10.1002/(SICI)1097-0134(19990601)35:4<408::AID-PROT4>3.0.CO;2-A. (原始內容存檔於2020-06-27).
- ^ 10.0 10.1 (英文)PDB Current Holdings Breakdown. [2008-01-08]. (原始內容存檔於2014-09-12).
- ^ Krimm, Samuel; Bandekar, J. Vibrational Spectroscopy and Conformation of Peptides, Polypeptides, and Proteins. Advances in Protein Chemistry. Advances in Protein Chemistry. 1986, 38 (C): 181–364. ISBN 9780120342389. doi:10.1016/S0065-3233(08)60528-8.
- ^ Lessing, J.; Roy, S.; Reppert, M.; Baer, M.; Marx, D.; Jansen, T.L.C.; Knoester, J.; Tokmakoff, A. Identifying Residual Structure in Intrinsically Disordered Systems: A 2D IR Spectroscopic Study of the GVGXPGVG Peptide. J. Am. Chem. Soc. 2012, 134: 5032–5035. doi:10.1021/ja2114135.
- ^ Jansen, T.L.C.; Knoester, J. Two-dimensional infrared population transfer spectroscopy for enhancing structural markers of proteins. Biophys. J. 2008, 94: 1818–1825. doi:10.1529/biophysj.107.118851.
- ^ (英文)Branden C, Tooze J. (1999). Introduction to Protein Structure 2nd ed. Garland Publishing: New York, NY
- ^ (英文)Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T. (2005). Lipid-protein interactions in double-layered two-dimensional AQP0 crystals. Nature 438(7068):633-8.
延伸閱讀
[編輯]- John Tooze, Introduction to Protein Structure, Garland, 1999, ISBN 0815323042
- 閻隆飛、孫之榮,《蛋白質分子結構》,清華大學出版社,1999年,ISBN 7302033293
外部連結
[編輯]- (英文) 如何利用貝葉斯算法從NMR資料進行結構計算 ── Habeck M, Nilges M, Rieping W. Bayesian inference applied to macromolecular structure determination (PDF). Physical review. E, Statistical, nonlinear, and soft matter physics. 2005, 72 (3 Pt 1): 031912 [2008-01-31]. PMID 16241487. (原始內容 (PDF)存檔於2007-09-30).
- (英文)ProSA-web (頁面存檔備份,存於網際網路檔案館) 查找實驗測定或理論計算所獲得的蛋白質結構中可能出現的錯誤的網絡伺服器
- (英文)NQ-Flipper 可以對蛋白質結構中Asn和Gln的異構物進行檢查的網絡伺服器
