核酸二级结构


核酸二级结构(英語:Nucleic acid secondary structure)是单个核酸聚合物内或两个聚合物之间的碱基对相互作用。它可以被表示为在核酸分子中配对的碱基的一个列表[1]。 生物DNA的和RNA的二级结构往往有所不同:生物DNA主要以完全碱基配对的双螺旋的形式存在,而生物RNA是单链的,并且由于来自核糖中额外的羟基增加的形成氢键的能力而形成错综复杂的碱基配对相互作用。
在非生物学背景下,由于碱基对的模式最终决定了分子的整体结构,因此二级结构对于DNA纳米技术和DNA运算的核酸结构的核酸设计至关重要。
基本概念
[编辑]碱基配对
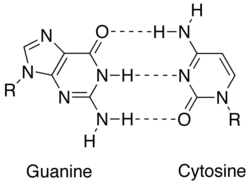
[编辑]在分子生物学中,通过氢键连接的互补DNA或RNA链上的两个核苷酸称为碱基对(通常缩写为bp)。在典型的沃森-克里克碱基配对中,DNA中的腺嘌呤(A) 与胸腺嘧啶(T)形成碱基对,鸟嘌呤(G)与胞嘧啶(C)形成碱基对。在RNA中,胸腺嘧啶被尿嘧啶(U)取代。交替氢键模式,如搖擺鹼基對和胡斯坦碱基对,也会出现——尤其是在RNA中——从而产生复杂且有功能的三级结构。重要的是,配对是蛋白质翻译过程中信使RNA分子上的密码子(codon)被转移RNA上的反密码子识别的机制。一些DNA或RNA结合酶可以识别特定的碱基配对模式,从而识别基因的特定调控区域。氢键是上述碱基配对规则背后的化学机制。氢键供体和受体的适当几何对应关系只允许“正确”的配对稳定形成。GC含量高的DNA比GC含量低的DNA更稳定,但与普遍看法相反,氢键并不能显著稳定DNA,稳定主要是由于堆积相互作用。[2]
较大的核碱基腺嘌呤和鸟嘌呤属于双环化学结构,称为嘌呤;较小的核碱基胞嘧啶和胸腺嘧啶(以及尿嘧啶)属于单环化学结构,称为嘧啶。嘌呤只能与嘧啶互补:嘧啶-嘧啶配对在能量上是不利的,因为分子之间相距太远,无法建立氢键;嘌呤-嘌呤配对在能量上是不利的,因为分子之间相距太近,导致重叠排斥。唯一可能的配对是GT和AC;这些配对属于错配,因为氢供体和受体的模式不一致。在RNA中,有两个氢键的GU搖擺鹼基對确实经常出现。
二级结构模体
[编辑]
核酸二级结构通常分为螺旋(连续碱基对)和各种环(螺旋包围未配对的核苷酸)。 通常将这些元素或它们的组合进一步分类为其他类别,包括例如四元环(Tetraloop),假结结构和茎环。
双螺旋
[编辑]双股螺旋是核酸分子中重要的核酸三级结构,与分子的二级结构紧密相连。双股螺旋由许多连续碱基对的区域形成。
核酸双螺旋是螺旋聚合物,通常是右旋的,含有两个碱基对配对的核苷酸链。单螺旋圈构成约十个核苷酸,并且包含大沟和小沟,大沟比小沟宽[3]。鉴于大沟和小沟宽度的差异,许多与DNA结合的蛋白质通过侵入较宽的大沟来实现[4]。对于DNA而言,许多双螺旋形式在理论上是可能的,三种生物中存在的形式是A-DNA,B-DNA和Z-DNA,而RNA双链只能形成与A形DNA相似的A形双螺结构旋。
茎环结构
[编辑]
核酸分子的二级结构通常可以独特地分解成茎(stem)和环(loop)。 茎环结构(通常也称为“发夹(hairpin)”),其中碱基配对的螺旋终止于短的不配对环中,是非常常见的,并且是用于较大结构基序如三叶草结构的构建模块,是四个螺旋结的连接,如转运RNA中被发现的那些。 内部环(较长的配对螺旋中的一系列不配对的碱基)和突起(螺旋的一条链具有“额外”插入的碱基而在相对链中没有对应物的区域)也是常见的。
假结结构
[编辑]
一个假结结构是含有至少两个茎环结构的核酸二级结构,其中一个茎的一半嵌入另一茎的两半之间。假结结构折叠成节状三维构象,但不是真正的拓扑结。 假结构中的碱基对不能很好地嵌套; 也就是说,碱基对出现在序列位置上彼此“重叠”。这使得核酸序列中一般假结的出现无法通过动态规划的标准方法来预测,该方法使用递归评分系统来识别配对的茎,并因此不能使用常用算法检测非嵌套碱基对。 然而,使用修改后的动态程序可以预测 有限的假结点的子类[6]。 较新的结构预测技术,如随机上下文无关文法也无法考虑假结结构。
二级结构预测
[编辑]大多数核酸二级结构预测方法都依赖于最近邻热力学模型。[7][8]给定核苷酸序列,确定最可能结构的常用方法是使用动态规划算法,该算法寻求寻找具有低自由能的结构。[9] 动态规划算法通常禁止假結結構或其他碱基对未完全嵌套的情况,因为即使对于较小的核酸分子,考虑这些结构的计算成本也非常高。其他方法,如随机上下文无关文法,也可用于预测核酸二级结构。
对于许多RNA分子而言,二级结构对于RNA的正确功能非常重要 — 通常比实际序列更重要。这一事实有助于分析有时称为“RNA基因”的非编码RNA。生物信息学的一个应用是使用预测的RNA二级结构在基因组中搜索非编码但有功能的RNA形式。例如,microRNA具有典型的长茎环结构,被小的内部环打断。
RNA二级结构适用于某些物种的RNA剪接。研究表明,在人类和其他四足动物中,如果没有U2AF2蛋白,剪接过程就会受到抑制。然而,在斑马鱼和其他真骨类鱼类中,即使没有U2AF2,某些基因上仍会发生RNA剪接过程。这可能是因为斑马鱼中10%的基因在每个内含子的3'剪接位点 (3'ss) 和5'剪接位点 (5'ss) 上分别有交替的TG和AC碱基对,从而改变了RNA的二级结构。这表明RNA的二级结构可以影响剪接,可能无需使用U2AF2等被认为是剪接发生所必需的蛋白质。[10]
二级结构测定
[编辑]RNA二级结构可以通过X射线晶体学获得的原子坐标(三级结构)来确定,这些坐标通常存储在蛋白质数据库中。目前的方法包括3DNA/DSSR[11] 和 MC-annotate[12]。
参阅
[编辑]参考资料
[编辑]- ^ Dirks, Robert M.; Lin, Milo; Winfree, Erik & Pierce, Niles A. Paradigms for computational nucleic acid design. Nucleic Acids Research. 2004, 32 (4): 1392–1403. PMC 390280
 . PMID 14990744. doi:10.1093/nar/gkh291.
. PMID 14990744. doi:10.1093/nar/gkh291.
- ^ Yakovchuk, Peter; Protozanova, Ekaterina; Frank-Kamenetskii, Maxim D. Base-stacking and base-pairing contributions into thermal stability of the DNA double helix. Nucleic Acids Research. 2006, 34 (2): 564–574. PMC 1360284
 . PMID 16449200. doi:10.1093/nar/gkj454.
. PMID 16449200. doi:10.1093/nar/gkj454.
- ^ Alberts; et al. The Molecular Biology of the Cell. New York: Garland Science. 1994. ISBN 978-0-8153-4105-5.
- ^ Pabo C, Sauer R. Protein-DNA recognition. Annu Rev Biochem. 1984, 53: 293–321. PMID 6236744. doi:10.1146/annurev.bi.53.070184.001453.
- ^ Chen JL, Greider CW. Functional analysis of the pseudoknot structure in human telomerase RNA. Proc Natl Acad Sci USA. 2005, 102 (23): 8080–5. PMC 1149427
 . PMID 15849264. doi:10.1073/pnas.0502259102.
. PMID 15849264. doi:10.1073/pnas.0502259102.
- ^ Rivas E, Eddy SR. A dynamic programming algorithm for RNA structure prediction including pseudoknots. J Mol Biol. 1999, 285: 2053–2068. PMID 9925784. doi:10.1006/jmbi.1998.2436.
- ^ Xia T, SantaLucia J Jr, Burkard ME, Kierzek R, Schroeder SJ, Jiao X, Cox C, Turner DH. Thermodynamic parameters for an expanded nearest-neighbor model for formation of RNA duplexes with Watson-Crick base pairs. Biochemistry. October 1998, 37 (42): 14719–35. CiteSeerX 10.1.1.579.6653
 . PMID 9778347. doi:10.1021/bi9809425.
. PMID 9778347. doi:10.1021/bi9809425.
- ^ Mathews DH, Disney MD, Childs JL, Schroeder SJ, Zuker M, Turner DH. Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure. PNAS. May 2004, 101 (19): 7287–92. Bibcode:2004PNAS..101.7287M. PMC 409911
 . PMID 15123812. doi:10.1073/pnas.0401799101
. PMID 15123812. doi:10.1073/pnas.0401799101  .
.
- ^ Zuker, M. On finding all suboptimal foldings of an RNA molecule. Science. 1989-04-07, 244 (4900): 48–52. Bibcode:1989Sci...244...48Z. ISSN 0036-8075. PMID 2468181. doi:10.1126/science.2468181 (英语).
- ^ Lin, Chien-Ling; Taggart, Allison J.; Lim, Kian Huat; Cygan, Kamil J.; Ferraris, Luciana; Creton, Robert; Huang, Yen-Tsung; Fairbrother, William G. RNA structure replaces the need for U2AF2 in splicing. Genome Research. 13 November 2015, 26 (1): 12–23. PMC 4691745
 . PMID 26566657. doi:10.1101/gr.181008.114.
. PMID 26566657. doi:10.1101/gr.181008.114.
- ^ Lu, XJ; Bussemaker, HJ; Olson, WK. DSSR: an integrated software tool for dissecting the spatial structure of RNA.. Nucleic Acids Research. 2 December 2015, 43 (21): e142. PMC 4666379
 . PMID 26184874. doi:10.1093/nar/gkv716.
. PMID 26184874. doi:10.1093/nar/gkv716.
- ^ MC-Annotate. www-lbit.iro.umontreal.ca.
外部链接
[编辑]- (英文)MDDNA: Structural Bioinformatics of DNA
- (英文)Abalone (页面存档备份,存于互联网档案馆) — Commercial software for DNA modeling
- (英文)DNAlive: a web interface to compute DNA physical properties. Also allows cross-linking of the results with the UCSC Genome browser and DNA dynamics.